Basic Operation of Jmol
Mouse Operation | Schemes | Measurement | Slab (Cutting Plane) | Reference Web address
マウスの機能 | モデルの種類の変更 | 距離と角度の表示 | Slab機能(断面の表示) | その他の情報の所在
Mouse Operation
Every action cna be achieved with one, two and three button mouse.
| Function | Main Button (Left) | Middle Button | Secondary Button (Right) |
|---|---|---|---|
| Open Jmol Menu | click on Jmol logo or hold Ctrl while clicking | click | |
| Rotate around X, Y | drag | ||
| Move along X,Y | hold Shift while double-clicking and holding down on the second click for dragging | double-click and holding down on the second click for dragging | hold Ctrl while dragging |
| Rotate around Z | hold Shift while dragging horizontally | drag horizontally | hold Shift while dragging horizontally (may fall on Mac's) |
| Zoom | hold Shift while dragging vertically or use mouse wheel |
drag vertically | |
| Reset | hold Shift while double-clicking away from the molecule | double-click away from the molecule |
Selection of Schemes
Radio Button Controls
You can use the radio button controls shown at the foot of the applet window.
Atoms are rendered as spheres based at the size of van der Waals radius of each element. "Off" corresponds to "Stick Model", "20%"corresponds to "Ball and Stick Model" and "100%" corresponds to "CPK (Spacefill) Model".
Jmol Menu
Jmol menu offers a wide assortment of options. Scheme is set using Style menu.
Style > Scheme > CPK Spacefill (Ball and Stick, Sticks, Wireframe)
In the case of proteins, the menu to use is Structures.
Style > Structures > Backbone (Off, Cartoon, Cartoon Rockets, Ribbons, Rockets, Strand, Trace)
 |
 |
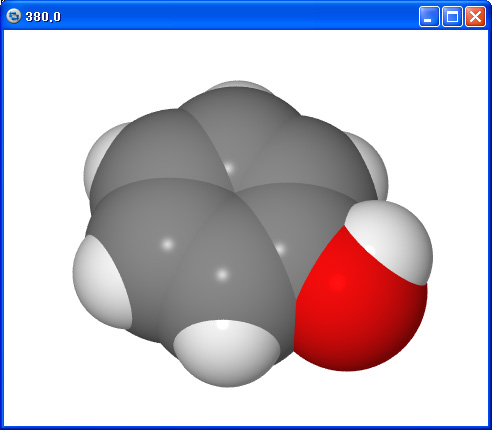
CPK model of phenol (High definition image produced by POV-Ray) |
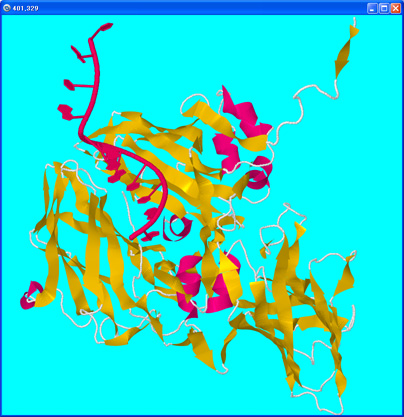
Protein drawn with Cartoon option |
Measurement of Distances and Angles
-
Distance (2 atoms):
- double-click on the starting atom
- to fix a distance measurement, double-click on second atom
-
Angle (3 atoms):
- double-click on the starting atom
- click on the second atom (central atom in angle)
- to fix an angle measurement, double-click on third atom
-
Torsion angle or dihedral (4 atoms)
- double-click on the starting atom
- click on the second atom
- click on the third atom
- to fix a dihedral angle measurement, double-click on fourth atom
- move outside the window in order to cancel the measurement
- make the same measurement again in order to delete the measurement
Slab - Cutting plane
Open the script dditor window. Type "slab on" and click the "Run" button.
| Function | Main button (Left) |
|---|---|
| Slab (slab from front) | hold Ctrl + Shift while dragging vertically |
| Depth (slab from back) | hold Ctrl + Shift while double-clicking and holding down on the second click for dragging vertically |
| Shift the slab | hold Alt + Ctrl + Shift while dragging vertically |
Example
 |
 |
CPK model of phenol (High definition image produced by POV-Ray) |
Cross section of phenol |
Reference Web address
Refernce guide for Jmol command and scripting language : http://chemapps.stolaf.edu/jmol/docs/?ver=11.0
Jmol web site : http://jmol.sourceforge.net
Jmol Applet の操作について
■ マウスの機能
Jmol では、 1 ボタン(例 Mac)、2 ボタン (例 Windows)、3 ボタン (例 Linux)及びホイールマウスが利用できます。
| 機 能 | 左(主)ボタン | 中央ボタン | 右ボタン |
|---|---|---|---|
| Jmol メニューの表示 | 右下のJmol のロゴをクリック または Ctrl +クリック |
クリック | |
| X,Y軸の回りの回転 | クリック & ドラッグ | ||
| X,Y方向の移動 | Shift+ダブルクリックした後、そのままドラッグ | ダブルクリックした後、そのままドラッグ | Ctrl+ クリックした後、そのままドラッグ |
| Z軸の回りの回転 | Shift+クリックした後、 水平方向に ドラッグ |
クリックした後、 水平方向にドラッグ | Shift+クリックした後、水平方向に ドラッグ (Macでは利用できない) |
| 拡大・縮小 | Shift+クリックした後、 垂直方向に ドラッグ または ホイールを回転 |
クリックした後、 垂直方向にドラッグ または ホイールを回転 |
|
| リセット | 分子の上以外で、Shift+ダブル クリック |
分子の上以外で、ダブルクリック |
■ 分子モデルの種類の変更
ラジオボタンなどによるContorolの利用
Appletウィンドウの下部に、ラジオボタンなどによるContorolが表示されている場合はそれを利用します。
Off が"Sticks" 、20%が "Ball and Stick"、100%が "CPKSpacefill"に相当します。
メニューの利用
Jmol メニューから、Style - Scheme を選び、"CPK Spacefill", "Ball and Stick","Sticks","Wireframe","Cartoon","Trace"のうちのいずれかを指定します。
タンパク質などの場合は、Style - Structures を選び、"Off","Backbone", "Cartoon","Cartoon Rockets","Ribbons","Rockets","Strand","Trace"のうちのいずれかを指定することができます。
 |
 |
PhenolのCPKモデルをPOV-Rayで出力した画像 |
タンパク質のCartoon表示 |
■ 距離と角度の測定
測定結果を残さずに値を表示させる場合
-
2 原子間の距離 :
-
最初の原子をダブルクリックします。
-
目的原子の上にマウスカーソルを移動させます。
-
-
3 原子間の角度 (結合角)
-
最初の原子をダブルクリックします。
-
2番目の原子をクリックします。
-
3番目の原子の上にマウスカーソルを移動させます。
-
-
4 原子間の角度 (二面角) :
-
最初の原子をダブルクリックします。
-
2番目の原子をクリックします。
-
3番目の原子をクリックします。
-
4 番目の原子の上にマウスカーソルを移動させます。
-
- 測定の途中でキャンセルするには、マウスカーソルをウィンドウの枠外に移動します。
測定結果を残す場合
-
2 原子間の距離 :
-
最初の原子をダブルクリックします。
-
2番目の原子をダブルクリックします。
-
-
3 原子間の角度 (結合角)
-
最初の原子をダブルクリックします。
-
2番目の 原子をクリックします。
-
3番目の 原子をダブルクリックします。
-
-
4 原子間の角度 (二面角) :
-
最初の原子をダブルクリックします。
-
2番目の 原子をクリックします。
-
3番目の 原子をクリックします。
-
4 番目の 原子をダブルクリックします。
-
測定値の表示を消すには、Jmolのウィンドウを再表示してください。
■ Slab(厚切り)機能 − 断面の表示
Script Editorのウインドウを開き、slab on と入力し、Run ボタンをクリックします。
| 機 能 | 左(主)ボタン |
|---|---|
| 前から | Ctrl + Shift キーを押した状態で垂直方向にドラッグする |
| 後から | Ctrl + Shift キーを押した状態でダブルクリックし、そのまま垂直方向にドラッグする |
| Slabを移動する | Alt + Ctrl + Shift キーを押した状態で垂直方向にドラッグする |
Slab機能を使用した例を示します。
 |
 |
PhenolのCPKモデルをPOV-Rayで出力した画像 |
PhenolのCPKモデルをSlab機能を使用して表示した画像 |
■ その他の情報
コンソール・ウィンドウで利用できるスクリプトに関しては、右記を参照してください : http://chemapps.stolaf.edu/jmol/docs/?ver=11.0
日本語による主なスクリプトに関する説明については、右記を参照してください : http://www3.u-toyama.ac.jp/kihara/soft/jmol/jmolscript.html
Jmolに関する詳しい情報については右記を参照してください : http://jmol.sourceforge.net