構文 [Jmol command syntax]
[Jmol math] − さまざまな変数と演算が利用可能です。
[Jmol parameters] − パラメータの値をセットして利用することができます。Set も参照してください。
[atom expressions] − キーワードや論理演算子を組み合わせて、条件に当てはまる原子の集合を指定することができます。
[atom properties] − 分子データに含まれる原子の性質を選択したり情報を収集することができます。
コメント
| # | その行の終わりまでをコメントとして無視します |
| /* テキスト */ | 囲まれた範囲を無視します |
| /** ‥‥ **/ | /* テキスト */ を含むブロック全体をコメントとみなします |
| // | 行頭にある場合、その行全体をコメントとみなします。 |
[export] − Jmolアプリケーションで、シーンをMaya, POV-Ray及びVRMLの形式のファイルとして書き出すことができます。詳細は write を参照してください。
[fractional coordinates] − 座標を単位格子に対する比で表すことができます。
[functions]
[plane expressions] − 面を定義します。
[reading ZIP/JAR files] − ZIPやJAR形式で圧縮されたファイルの中の指定したファイルを読み込みます。
[status reporting] − Appletの状態を報告します。
[using the clipboard]
アニメーション animation
アニメーションのパラメータを指定したり、アニメーションの開始/終了を指定します。データファイル中の複数の構造を順次表示する場合のアニメーションのみを制御します。
| animation ON/OFF {default: ON} | アニメーションの開始/終了を指定します |
| animation direction -1 | アニメーションの方向を逆向きにします。(最後のフレームから最初のフレームの向き) |
| animation direction 1 | アニメーションの方向を正方向にします。(最初のフレームから最後のフレームの向き) |
| animation fps 整数 | アニメーションの速さ(1秒当りのフレーム数)を指定します |
| animation frame | frame を参照してください |
| animation mode LOOP | アニメーションを自動的に繰り返します |
| animation mode LOOP t1 t2 | アニメーションの始めと終わりでそれぞれt1秒とt2秒だけ待ってアニメーションを自動的に繰り返します |
| animation mode ONCE | アニメーションを一度だけ実行します(デフォルト) |
| animation mode PALINDROME | アニメーションを振り子状に自動的に繰り返します |
| animation mode PALINDROME t1 t2 | アニメーションの始めと終わりでそれぞれt1秒とt2秒だけ待ってアニメーションを振り子状に自動的に繰り返します |
座標軸の表示 axes
| axes ON/OFF {default: ON} | 座標軸の表示/非表示を指定します |
| axes [直径] | 座標軸の太さ(直径)を指定します。実数の場合はÅ単位、整数の場合はピクセル単位となります。 |
| axes DOTTED | 座標軸を細い点線で表示します |
| axes MOLECULAR | 座標軸を細い点線で表示します |
| axes SCALE スケール | 座標軸の長さを指定します |
| axes POSITION [x y] or [x y %] | ウィンドウ内の指定した位置にもとづいて座標軸を設定します。 |
| axes UNITCELL | 単位格子のa, b, c軸に沿って座標軸を設定します。(分数座標の場合のデフォルト) |
| axes WINDOW | バウンディングボックスの中心にもとづいて座標軸を設定します。(分数座標でない場合のデフォルト) |
骨格の表示 backbone
タンパク質や核酸のα炭素を結ぶ骨格を表示します。
| backbone ON/OFF {default: ON} | 骨格の表示/非表示を指定します |
| backbone ONLY | 骨格のみを表示します。 |
| backbone 半径 | 骨格の半径をÅ単位で指定します。(4Å以下) |
背景の指定 background
| background RGBカラー | 背景色を指定します。RGBカラーについては、Jmolで使われている色について を参照してください。 |
| background BIND マウス操作 jmol動作 | マウス操作をJmolの動作に関連づけます。 |
| background ECHO 色 | テキストの背景色を指定します。色については、Jmolで使われている色について を参照してください。 |
| background IMAGE "ファイル名" | 指定したイメージファイルを背景として指定します。JPG, PNGまたはGIFが利用できます。 |
| background BIND マウス操作 スクリプト | マウス操作をJmolのスクリプトに関連づけます。 |
| background HOVER [color-none-CPK] | マウスカーソルの位置に表示されるテキストの背景色を指定します。色については、Jmolで使われている色について を参照してください。 |
| background LABELS 色 | ラベルの背景色を指定します。色については、Jmolで使われている色について を参照してください。 |
結合多重度の指定 bondorder
選択されている原子や結合の多重度を指定します。connect も参照してください。
| bondorder 多重度 | 多重度 0.5, 1, 1.5, 2, 2.5, 3, 4, -1, -1.5, -2.5 を指定します。-1 は HBOND, -1.5 はPARTIALDOUBLE, -2.5 は PARTIALTRIPLE2に相当します。 |
| bondorder 多重度の型 | SINGLE, DOUBLE, TRIPLE, QUADRUPLE, AROMATIC, PARTIAL, PARTIALDOUBLE, PARTIALTRIPLE, PARTIALTRIPLE2, PARTIAL N.M, UNSPECIFIEDまたは HBONDのうちから指定します。PARTIAL N.Mについてはconnect を参照してください。 |
カーツーン cartoon
αへリックス、βシートなどをモデル化した形で表示します。
| cartoon ON/OFF {default: ON} | αへリックス、βシートなどをモデル化した形で表示するかしないかを指定します |
| cartoon ONLY | Cartoon のみを表示し、そのほかを表示しません。 |
| cartoon 半径 | Cartoonの半径をÅ単位で指定します。(4Å以下) |
boundbox
calculate −
cd − デフォルトディレクトリを変更します。またはデフォルトディレクトリを表示します。
中心の指定 center
分子の中心を移動させます。分子の中心は回転の中心になります。
| center 原子範囲の指定 | 指定した範囲に含まれる原子の中心を分子の中心とします。 |
| center XYZ座標 | 指定した座標を分子の中心とします。 |
| center 図形 | 指定した図形の中心を分子の中心とします。 |
| center | デフォルト位置に戻します |
centerAt
color
color (atom object)
color (bond object)
color (element)
color (model object)
color (named object)
color (scheme)
color measures
color selectionHalos
configuration
connect
console − Jmol Script Console ウィンドウを表示します。
data − 原子のプロパティを設定します。
define − 選択された原子に対してグループ名を定義します。
delay − 画面を書き換え、指定した時間だけ実行を停止します。
depth − 断面表示での表示開始位置を指定します。slabの項を参照してください。
dipole − 双極子を示す矢印を描きます。
display − 指定した原子またはグループのみを表示します。
dots
draw
echo
ellipsoid
exit
font
for
frame
frank
geoSurface
getProperty
goto
halos
hbonds
help
hide
history
hover
if_while_for
initialize
invertSelected
等高面の表示 isosurface
isosurface [object id] [construction/mapping parameters] [surface object] [additional mapping-only parameters] MAP [color mapping dataset] [display options]
[construction/mapping parameters]-- molecular/solvent surfaces
| ADDHYDROGENS | solvent または sasurface objectについて、,面を生成する前に不足している水素原子を付ける |
| IGNORE {atom expression} | Specifies a set of atoms to completely ignore when generating a solvent-related surface (SOLVENT or SASURFACE). Typically these might be solvent molecules or atoms: IGNORE {solvent}. |
| IONIC radius | 次の番号の分子軌道を表示する |
| SELECT {atom_exp} | 一つ前の番号の分子軌道を表示する |
| SET n | HOMOまたはHOMOから+/-n番目の分子軌道を表示するかを指定する |
| VDW radius | LUMOまたは LUMOから+/-n番目の分子軌道を表示するかを指定する |
| WITHIN x.xx {atomExpression or point} | 分子軌道を消去する(どの分子軌道を表示するかの指定を削除する) |
[]
javascript
label
lcaoCartoon
load − ファイルの読み込み
- 書式
load "filename"
Jmolでは、ファイルの内容によってタイプを判断しているので、拡張子は何でもかまいません。
Jmolがサポートしているファイルフォーマットを参照してください。
load APPEND − 現在のモデルを置き換えることなく、モデルを追加します。
load FILES − 複数のファイルを読み込みます。
load MENU − 独自のメニューを読み込みます。
load MODELS − 複数のモデルの情報を含むファイルを読み込みます。
load TRAJECTORY − ファイルを trajectory として読み込みます。
load [property] − 指定した性質の情報のみを読み込んで、選択されている原子に適用します。
loop − スクリプトを繰り返し実行します。
measure
meshribbon
message
minimize
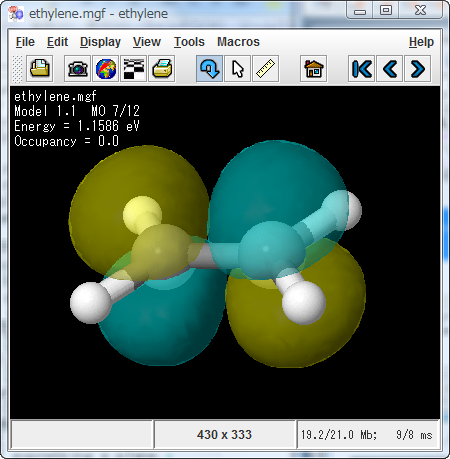
分子軌道の表示 mo
| mo ON/OFF | 分子軌道を表示する/しない {default: ON} |
| mo (整数) | 何番目の分子軌道を表示するかを指定する |
| mo NEXT | 次の番号の分子軌道を表示する |
| mo PREVIOUS | 一つ前の番号の分子軌道を表示する |
| mo HOMO [+/-n] | HOMOまたはHOMOから+/-n番目の分子軌道を表示するかを指定する |
| mo LUMO [+/-n] | LUMOまたは LUMOから+/-n番目の分子軌道を表示するかを指定する |
| mo DELETE | 分子軌道を消去する(どの分子軌道を表示するかの指定を削除する) |
| mo [mesh/nomesh] | 格子あるいは面で表示するかを指定する {default: mesh} |
| mo [fill/nofill] | 格子内(面)を塗りつぶすか否かを指定する {default: nofill} |
| mo [translucent/opaque] | 半透明/不透明を指定する {default: opaque} |
| mo COLOR 色 | 軌道のローブを描く色を指定する。色は、color name、 [r, g, b]または[xRRGGBB]の形式で指定する。例 mo color magenta、mo color [255,0,255]、mo color [xFF00FF] |
| mo COLOR 色1 色2 | 波動関数の値が負の部分と正の部分を描く色をそれぞれ指定する色は、color name、 [r, g, b]または[xRRGGBB]の形式で指定する。 |
| mo CUTOFF (実数l) | 軌道の等値面のカットオフ値を指定する。0.005 - 0.05の範囲で試してみて適切な値を用いると良い。 符号なしの正の値により正負の等値面のカットオフ値を指定する。+/-の符号を付けた場合は、それぞれ正負の等値面のみを指定することになる。 |
| mo RESOLUTION (実数l) | 等値面を作成する際のオングストローム当たりの格子点の数を指定する。値を大きくすると、拡大した場合にも等値面が滑らかに表示されるが、表示に時間が掛かることになる。 例 mo resolution 6 |
| mo PLANE plane_expression | 指定した面での断面を表示する。 例 mo plane "xy" mo plane {1 1 1 0} {a b c d} の形で、ax + by + cz = dの式で表される面を指定する。 |
| mo NOPLANE | 断面ではなく通常の表示に戻す |
| axes ON/OFF | 座標軸を表示する {default: OFF} |
| mo TITLEFORMAT "format" | タイトルのフォーマットを指定する。%E (エネルギー), %F (ファイル名), %I (MOの番号), %M (モデルの番号), %N (MOの数), %O (占有数), %S (対称性), and %U (エネルギー単位) | (改行) ? (データがなければ無視する) デフォルトは、 "%F|Model %M MO %I/%N|Energy = %E %U|?Symmetry = %S|?Occupancy = %O" |
分子軌道の表示例
エチレンのLUMO
スクリプト mo lumo; mo fill nomesh; mo translucent; mo color yellow cyan